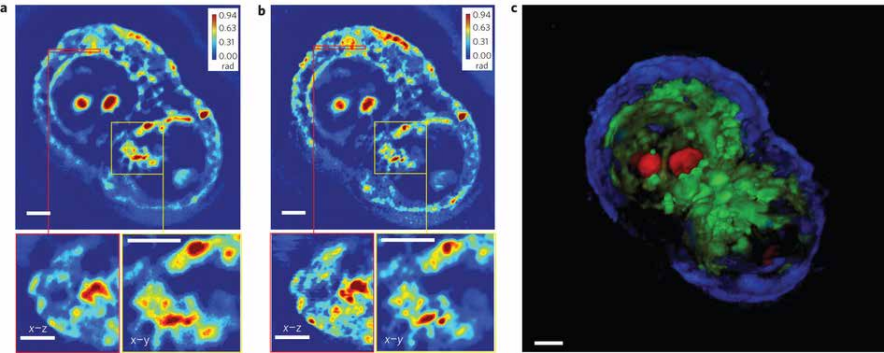
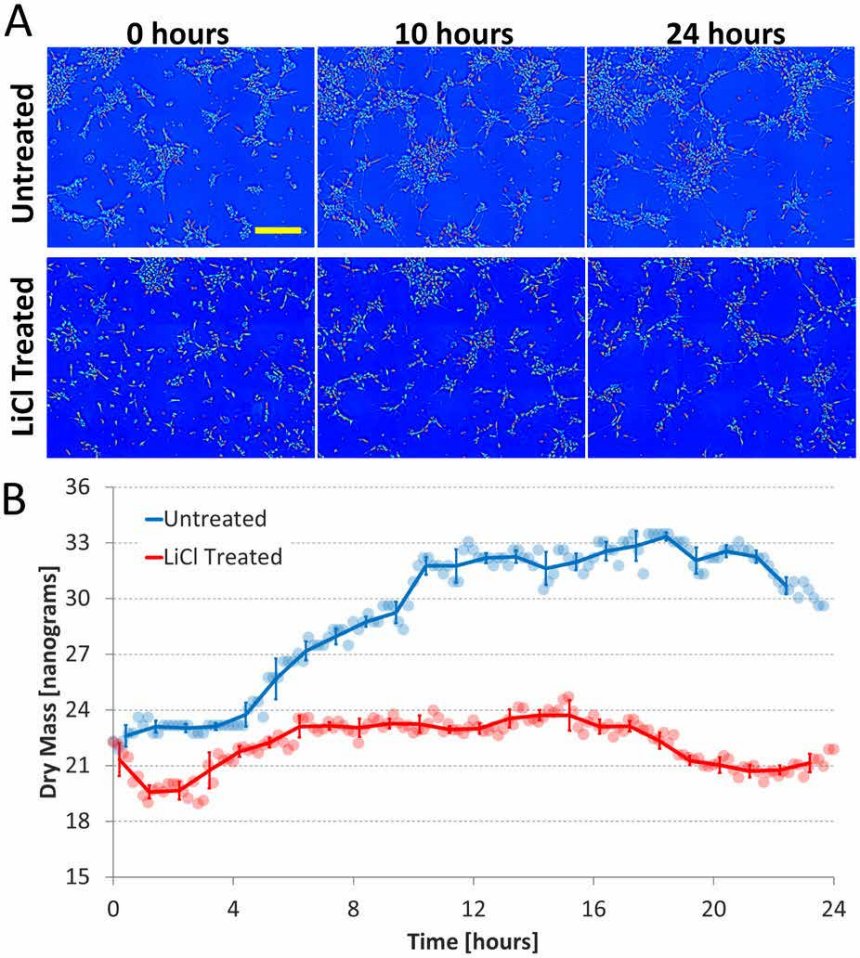
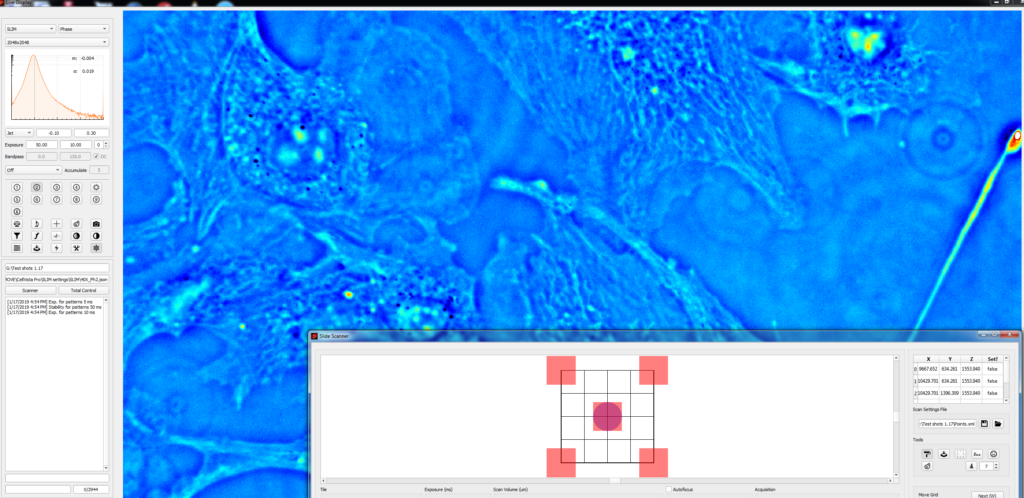
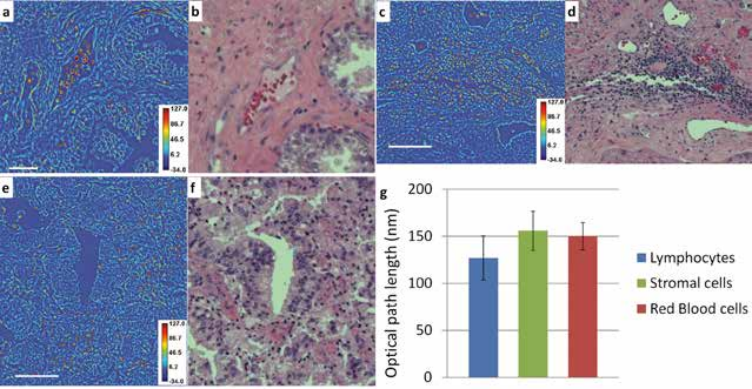
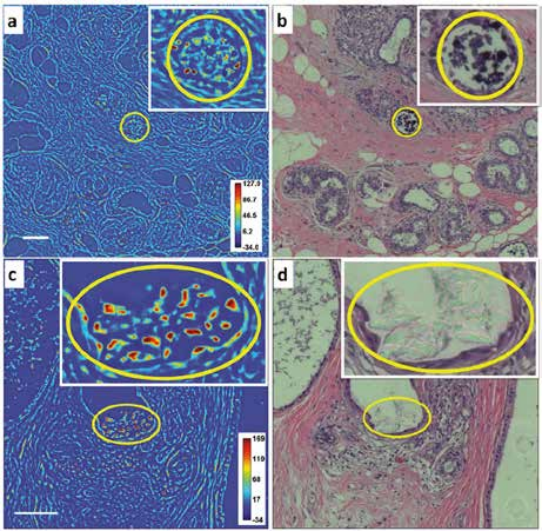
Технология SLIM™- это не инвазивный метод количественно-фазовой визуализации, который количественно определяет физические свойства живых клеток и тканей. В результате анализа данных получается количественное изображение (SLIM™-карта) образца в режиме реального времени. Интенсивность каждого пикселя в кадре является мерой карты фазового сдвига (в радианах) или разности длин оптического пути (в нанометрах) через образец, которая измеряется с чувствительностью более 0.5 нанометров [1]. Как показано на рис. 1, карта фазового сдвига конвертируется без задержки по времени в другие SLIM™-карты с соответствующей интенсивностью пикселей: толщиной (в микронах), плотностью сухой массы (пикограммы на квадратный микрон) и индексом преломления.
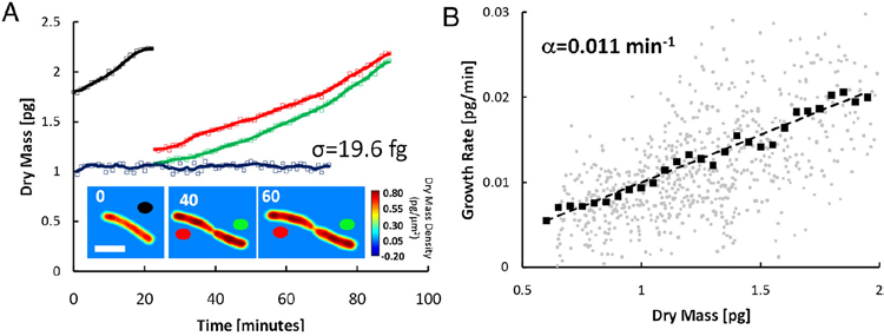
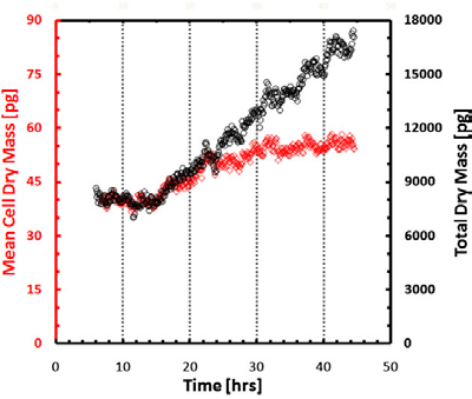
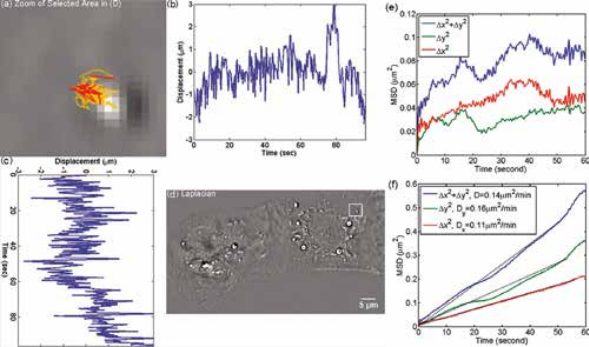
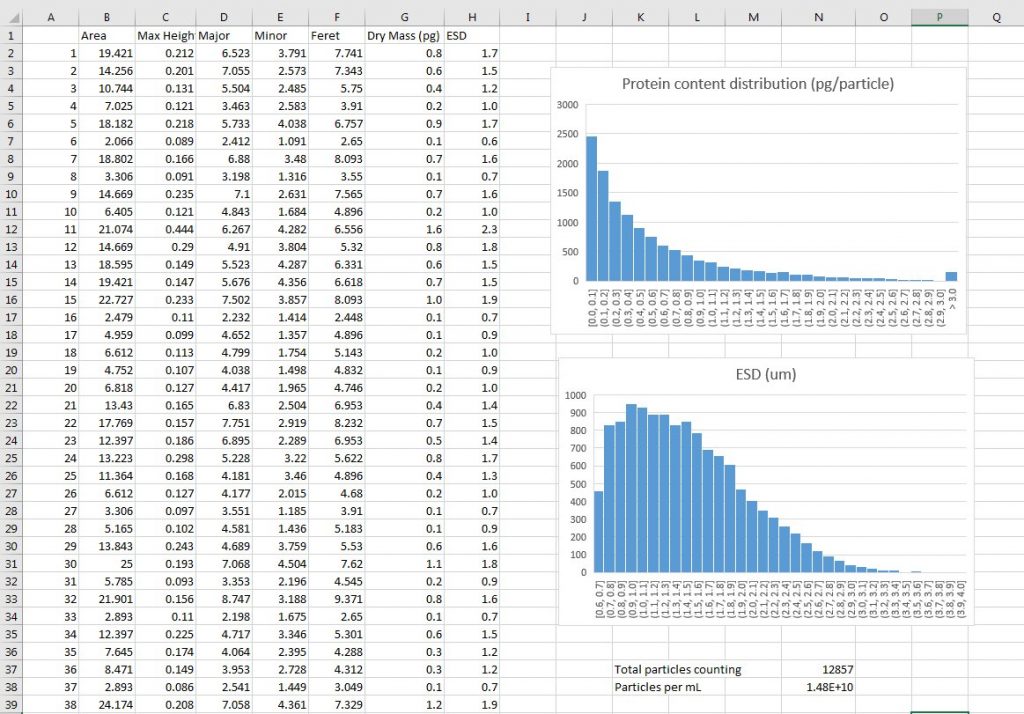
Технология SLIM™ была использована для измерения сухой массы отдельных клеток E-coli, как показано на рисунке 3. Измерения проводились одновременно на отдельных клетках, и соответствующая статистика была получена на больших популяциях.
SLIM™ измерения роста E. coli. (А) Сухая масса vs. времени для семейства клеток. Кривые роста для каждой клетки обозначаются цветными кружками на изображениях. На снимках показаны карты плотности сухой массы одной клетки в указанные моменты времени (в минутах). (Масштаб: 2 мкм.) Синяя линия — фиксированное измерение ячеек с SD 19,6 19.6 фемтограмм. Маркеры отображают необработанные данные, сплошные линии — усредненные данные. (B) Скорость роста vs. массы 20 клеток измеряется таким же образом. Слабые круги указывают на отдельные точки данных из отдельных кривых роста клеток, темные квадраты показывают среднее значение, а пунктирная линия — линейное соответствие усредненным данным; наклон этой линии, 0,011 мин-1, является мерой средней константы роста для данной популяции. Линейная зависимость между скоростью роста и массой указывает на то, что в среднем клетки E.coli демонстрируют экспоненциальное поведение роста [2].
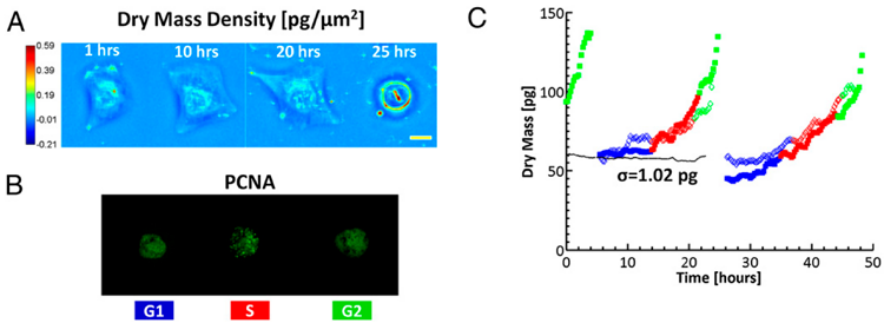
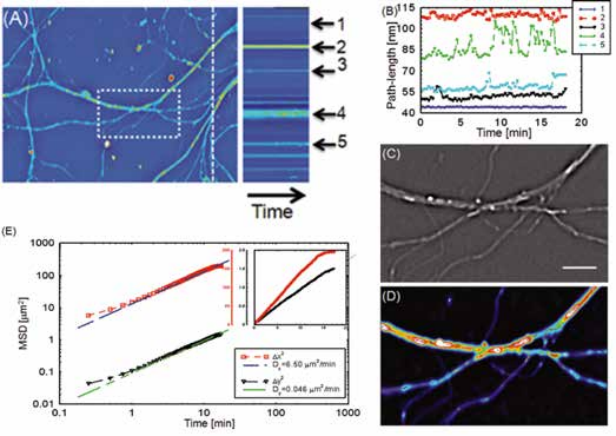
Для изучения роста одиночных клеток в асинхронной культуре и получения информации о росте, зависящем от цикла клеток, технологию SLIM™ использовали вместе с эпифлуоресцентной визуализацией, как показано на рис. 4 [2]. В то время как по технологии SLIM измерялась сухая масс в каждый момент времени, флуоресцентные маркеры указывали на фазу клетки в пределах клеточного цикла. Технология SLIM™ использует в микроскопе один и тот же оптический путь, что другими каналы, это обеспечивает бесшовное наложение всех обычных модальностей микроскопа.
Соответствие один к одному между клетками на изображениях SLIM™ и клетками на флуоресцентных изображениях позволяет проводить зависящие от цикла измерения роста. Сухие карты плотности массы (А) одной клетки U2OS на протяжении всего ее цикла в указанное время. (Масштаб: 25 мкм) Цветная линейка — плотность сухой массы в пг/мкм2. (В) Одновременное получение флуоресцентных изображений GFP, указывающих на активность PCNA; отчетливый сигнал GFP во время S-фазы и морфологические изменения во время митоза позволяют определить фазу цикла клетки. (С) Сухая масса vs время для семейства клеток (т. е. клетки 1->2->4). Две различные линии дочерних клеток дифференцируются по заполненным и открытым маркерам; для наглядности показана только одна дочерняя клетка от каждого родителя. Различные цвета указывают на цикл клетки, о котором сообщает флуоресценция GFP-PCNA. Черная пунктирная линия показывает измерения от неподвижной клетки, SD которой составляет 1,02 пг [2].
Результаты наложения сигнала флуоресценции на данные , полученные при помощи технологии SLIM™ (видео презентация).
Результаты наложения сигнала флуоресценции , меченных митохондрии белком GFP в клетках CHO-K1 (зеленый канал) на данные , полученные при помощи технологии SLIM™ (красный канал) в режиме реального времени.
2D-Оптическая визуализация в режиме реального времени бактерий при помощи технологии SLIM™, GLIM™ (видео презентация).
Публикации
[1] G. Popescu (2011) Quantitative phase imaging of cells and tissues (McGraw-Hill, New York) p. 313.
[2] M. Mir, Z. Wang, Z. Shen, M. Bednarz, R. Bashir, I. Golding, S. Prasanth and G.Popescu, Optical Measurement of cycle-dependent growth , Proc. Natl. Acad. Sci., 108 (32), 13124 (2011).
[3] M. Mir, T. Kim, A. Majumder, M. Xiang, R. Wang, S. C. Liu, M. U. Gillette, S. Stice and G. Popescu, Label-free characterization of emerging human neuronal networks, Scientific Reports, 4, 4434 (2014)
[4] M. Mir, A. Bergamaschi, B. S. Katzenellenbogen and G. Popescu, Highly sensitive quantitative imaging for monitoring single cancer cell growth kinetics and drug response, PLoS ONE, 9 (2), e89000 (2014)